18.01.2019
5 minutes de lecture
Le prix Yves Chauvin 2018 a été attribué par le conseil scientifique d’IFPEN à Aurélie Pirayre pour son travail de thèse intitulé « Reconstruction and Clustering with Graph optimization and Priors on Gene networks and Images ».
Aurélie Pirayre a reçu son prix lors de la cérémonie qui s'est tenue à IFPEN le 19 novembre 2018 à Rueil-Malmaison.
Ses travaux représentent une avancée dans l’adaptation de méthodes de traitement d’images à fin de représentation de données biologiques sous forme de graphes. Dirigés par Jean-Christophe Pesquet (Université Paris-Est Marne-la-Vallée, désormais CentraleSupélec), ils ont été encadrés au sein d’IFPEN par Frédérique Bidard-Michelot et Laurent Duval.
Après avoir soutenu sa thèse en juillet 2017, Aurélie Pirayre a rejoint les équipes de la direction Sciences et Technologies du numérique d’IFPEN.
Quel était le sujet de vos recherches ?
Mes recherches ont porté sur l’amélioration de la compréhension des mécanismes de production d’enzymes par un champignon, Trichoderma reesei. Ces enzymes spécifiques sont utilisées en tant que biocatalyseurs, pour dégrader la biomasse en sucres simples dans certains procédés de fabrication de bioéthanol de 2e génération. Mes travaux ont consisté à développer des approches mathématiques permettant de mieux connaître le fonctionnement génétique de ce champignon, dans le but d’améliorer le procédé de production des enzymes.
Les recherches ont été lancées en collaboration entre les départements « Biotechnologie » et « Contrôle, Signal et Système » d’IFPEN afin de tirer parti des compétences présentes dans les deux disciplines : en biologie et en mathématiques pour le traitement d’images et l’optimisation. Tout en initiant une thématique bio-informatique à IFPEN, elles ont généré une approche et des outils qui ont débouchés sur une meilleure exploitation des données biologiques.
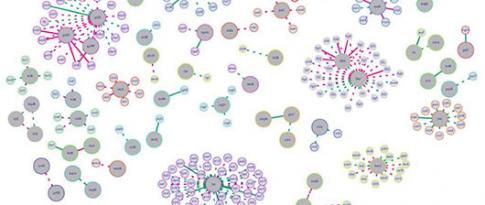
Ainsi, nous avons pu matérialiser, sous forme de graphes, les cascades de réactions entre les gènes qui décrivent le fonctionnement génétique de Trichoderma reesei. Les points du graphe correspondent aux gènes et les liens entre les points correspondent aux interactions entre les gènes. Afin d’obtenir des réseaux biologiquement réalistes, nous avons intégré des informations biologiques à ces méthodes de traitement d’images. C’est là que réside l’originalité de mes travaux : on a pris du savoir biologique que l’on a traduit en formules mathématiques, pour l’intégrer dans des modèles de traitement d’images existants.
A partir de cette approche, nous avons développé plusieurs outils capitalisés sous un nom générique : BRANE (Biologically-Related Apriori for Network Enhancement). Deux principaux outils, BRANE Cut et BRANE Clust, ont été publiés dans des revues scientifiques de bio-informatique de premier plan, ainsi que d’autres travaux dans des revues spécialisées en biologie et en mathématiques.
Ces recherches vont-elles être poursuivies ?
Le champignon Trichoderma reesei reste encore assez mal connu. Mes travaux ont permis de mettre en lumière des mécanismes régulateurs complexes pour la production de biocatalyseurs.
Les recherches vont se poursuivre dans le cadre de deux thèses qui vont s’attacher à la même problématique de fond, à savoir améliorer la compréhension sur le fonctionnement génétique de Trichoderma reesei. Une thèse sera plus axée vers les mathématiques, et l’autre vers la biologie. Ces deux thèses seront menées au sein de l’équipe bio-informatique.
Quant à la suite d’outils BRANE, à l’heure actuelle elle tend à être déployée sur de nouveaux projets portant sur d’autres micro-organismes étudiés à IFPEN. La suite BRANE a été développée de manière à pouvoir être utilisée par les biologistes au quotidien, avec leurs données. La bio-informatique est en plein essor, et de nouvelles technologies d’acquisition de la donnée biologique sont mises en place, ce qui génère énormément de données. On retrouve les problématiques liées à ce que l’on qualifie de « Big Data », avec de gros volumes de données hétérogènes, et c’est là que les mathématiques et l’informatique deviennent indispensables au biologiste afin qu’il puisse traiter ces données.
Qu’avez-vous apprécié dans le fait de faire votre thèse à IFPEN ?
IFPEN est un très bon endroit pour faire une thèse, les doctorants y sont choyés. Beaucoup de choses sont mises en œuvre pour qu’ils se sentent intégrés : des journées d’intégration et des séminaires thématiques par exemple. Les doctorants ont accès à un environnement scientifique riche en formations, de très bonne qualité, qui sont véritablement un plus pour ceux qui souhaitent continuer à travailler sur des thématiques liées à l’énergie et aux transports. Il y a également une association des doctorants, l’ADIFP, qui met en place des activités comme des cours d’anglais ou un voyage d’étude, et qui participe activement à l’organisation de divers événements comme par exemple la journée « Doc Day IFPEN », journée de rencontre avec des entreprises.
Lors de ma thèse, l’équipe encadrante a été très présente, très à l’écoute, d’autant plus qu’il s’agissait du premier sujet en bio-informatique à IFPEN. Il y avait des équipes spécialisées en mathématiques et en biologie, qui sont des domaines complètement différents n’utilisant pas le même vocabulaire. J’ai été choisie pour ma double compétence, qui m’a permis de faire le pont entre ces deux univers de manière assez fluide et rapidement. Il y a vraiment eu une écoute mutuelle pour bien cerner les problèmes et essayer de les résoudre ensemble. Cette cohésion d’équipe a été cruciale dans la réussite de mes recherches.
Publications
- A. Pirayre, C. Couprie, F. Bidard, L. Duval, J-C. Pesquet. BRANE Cut: biologically-related a priori network enhancement with graph cuts for gene regulatory network inference, BMC Bioinformatics, 2015.
>> DOI: 10.1186/s12859-015-0754-2
- A. Pirayre, C. Couprie, L. Duval, J-C. Pesquet. BRANE Clust: Cluster-Assisted Gene Regulatory Network Inference Refinement, IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2018, Vol. 15, Issue 3.
>> DOI: 10.1109/TCBB.2017.2688355
Vous serez aussi intéressé par
‘‘BRANE Power’’ : gènes et algorithmes, une alliance pour la chimie verte
THÈSE D’AURÉLIE PIRAYRE, PRIX YVES CHAUVIN 2018