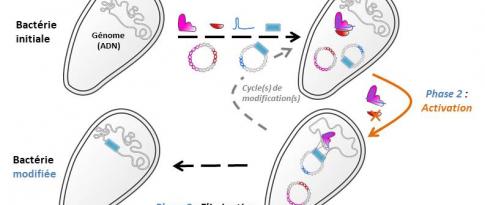
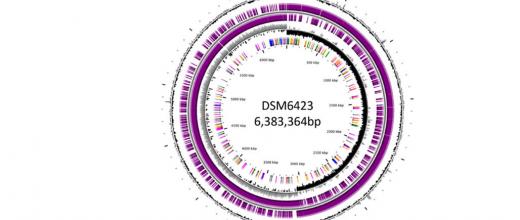
Avant leur mise en œuvre dans les procédés de production de biocarburants ou de molécules chimiques biosourcées, les micro-organismes impliqués doivent être optimisés. Cela passe par de l’ingénierie génétique qui consiste à inactiver ou ajouter un ou plusieurs gènes afin d’améliorer l’aptitude à la production de molécules cibles. L’identification des leviers d’amélioration demande une solide connaissance des systèmes biologiques complexes sur lesquels des progrès sont nécessaires.
L’explosion des techniques d’analyse « omiques » ces vingt dernières années a permis de caractériser et quantifier en globalité les molécules biologiques constitutives d’une cellule et de mieux appréhender son phénotype (les traits observables, par exemple la production d’une molécule d’intérêt). Désormais, plusieurs niveaux de données peuvent être collectés pour établir les relations entre les différentes macromolécules (ADN, ARN, protéines, métabolites) d’un système biologique (figure).
À IFPEN, le recours à la génomique (ADN) a contribué à rassembler des données sur la composition et la structure des génomes des micro-organismes utilisés au sein du département Biotechnologie [1,2]. Il nous est ainsi possible d’établir finement la cartographie génétique des souches industrielles produites puis de la lier aux phénotypes observés.
Cliquer sur l'image pour l'agrandir
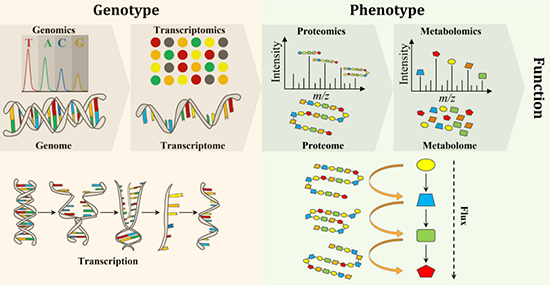
Les étapes suivantes d’analyse consistent à suivre l’expression des milliers de gènes d’un micro-organisme et la synthèse de leur produit, via des approches de transcriptomique (ARN), protéomique (protéines) et métabolomique (métabolites). Les outils développés par ces approches [4,5] sont aujourd’hui utilisés en routine, y compris pour des travaux de thèse, afin de répondre aux questions liées à l’optimisation de nos micro-organismes. Ils contribuent à affiner notre conduite des procédés fermentaires. Les résultats obtenus lors de la thèse de Rémi Hocq, récipiendaire pour ce travail de trois prix sur 2020-2021, sont principalement basés sur l’utilisation de ces outils.
Plus récemment, des méthodologies rassemblant des informations sur les régulations épigénétiquesa de l’expression des gènes ont été développées dans le cadre d’un autre travail doctoral. Des données d’épigénomique ont conduit à identifier des mécanismes de régulation inédits impliqués dans la production de cellulases dans Trichoderma reesei, un micro-organisme modèle impliqué dans nos procédés d’hydrolyse de la biomasse lignocellulosique [6].
Enfin, la recherche de nouvelles activités enzymatiques ou de nouveaux micro-organismes est réalisée via l’analyse d’écosystèmes complexes. Dans ce cas, des approches de métagénomique et/ou de métatranscriptomique sont employées. Ces approches permettent non seulement de définir la composition microbiologique d’un biotope donné – tel qu’un environnement pouvant abriter des micro-organismes d’intérêt ou des sous-sols liés à des projets d’énergie renouvelable (géothermie ou stockage de l’hydrogène) – mais aussi d’identifier les enzymes présentes et les voies de synthèse actives.
En conclusion, l’utilisation et la maîtrise des outils « omiques » à IFPEN ont un effet à double détente : approfondir nos connaissances sur les micro-organismes d’intérêt et optimiser ceux-ci pour les intégrer dans des bioprocédés innovants.
a- Discipline de la biologie qui étudie la nature des mécanismes modifiant de manière réversible, transmissible et adaptative l'expression des gènes sans en changer la séquence nucléotidique
Références :
- Jourdier, E., Baudry, L., Poggi-Parodi, D., Vicq, Y., Koszul, R., Margeot, A., Bidard, F. (2017). Proximity ligation scaffolding and comparison of two Trichoderma reesei strains genomes. Biotechnology for biofuels, 10 (1), 1-13.
>> doi.org/10.1186/s13068-017-0837-6
- Máté de Gérando, H. M., Wasels, F., Bisson, A., Clement, B., Bidard, F., Jourdier, E., … & Ferreira, N. L. (2018). Genome and transcriptome of the natural isopropanol producer Clostridium beijerinckii DSM6423. BMC genomics, 19 (1), 1-12.
>> doi.org/10.1186/s12864-018-4636-7
- Amer, B., & Baidoo, E. E. (2021). Omics-Driven Biotechnology for Industrial Applications. Frontiers in Bioengineering and Biotechnology, 9, 30.
>> doi.org/10.3389/fbioe.2021.613307
- Hocq, R., Jagtap, S., Boutard, M., Tolonen, A.C., Duval, L., Pirayre, A.,Lopes Ferreira, N., & Wasels, F. Genome-wide TSS distribution in three related Clostridia with normalized Capp-Switch sequencing. Microbiology Spectrum, e02288-21.
>> doi.org/10.1128/spectrum.02288-21
- Poggi-Parodi, D., Bidard, F., Pirayre, A., Portnoy, T., Blugeon, C., Seiboth, B., … & Margeot, A. (2014). Kinetic transcriptome analysis reveals an essentially intact induction system in a cellulase hyper-producer Trichoderma reesei strain. Biotechnology for biofuels, 7 (1), 1-16.
>> doi.org/10.1186/s13068-014-0173-z
- Fajon S., Jourdier E., Bidard F. (2021). Souche de champignon hyperproductrice de protéines. Brevet 21/11.274
Lexique
ADN : acide nucléique, support de l’information génétique
ARN : acide nucléique permettant la synthèse de protéines
Métabolite : substance organique intermédiaire ou issue du métabolisme
Epigénétique : étude de la nature des mécanismes modifiant l’expression des gènes sans en modifier la séquence
Métagénome : ensemble des gènes contenus dans un biotope donné, toutes espèces mélangées
Métatranscriptome : ensemble des gènes exprimés dans un biotope donné, toutes espèces mélangées
Cellulase : biocatalyseur capable de dégrader la cellulose (biopolymère)
Contacts scientifiques : Frédérique Bidard-Michelot, Francois Wasels
Vous serez aussi intéressé par
Hydrolyse de la biomasse lignocellulosique : étude des interactions enzyme-substrat (HDR 2015)
Le périmètre de mon HDR a couvert dix années de recherche à IFPEN dans le contexte du développement de Futurol™, procédé destiné à produire du bioéthanol de 2e g
‘‘BRANE Power’’ : gènes et algorithmes, une alliance pour la chimie verte
THÈSE D’AURÉLIE PIRAYRE, PRIX YVES CHAUVIN 2018