06.12.2023
4 minutes de lecture
Grâce au génie génétique, les chercheurs comprennent de mieux en mieux les processus du vivant et injectent cette connaissance dans des biotechnologies à vocation industrielle, pour décarboner l'énergie et la chimie. Dans ce cadre, les équipes d’IFPEN en biotechnologies mènent des travaux pour améliorer l’efficacité des enzymes dans la transformation de la cellulose et autres polysaccharides des plantes en sucres fermentescibles. Ces travaux ont ainsi contribué à faire du procédé Futurol™ une technologie de pointe promise à un bel avenir. Pour progresser dans cette compréhension globale du vivant, ils font aussi appel désormais à des traitements algorithmiques complexes permettant d’interpréter les données éparses et incomplètes issues de l’expérimentation. Une analogie est ici proposée pour planter le décor et explorer ce monde nouveau.

En quête du vivant : l’audience est ouverte
Dans un tribunal, un procès à huis-clos se tient. Peut-on, sans y avoir assisté, comprendre les éléments qui ont mené au verdict rendu ? Nous ne disposons qu’a posteriori d’informations partielles sur ce qu’il s’est passé durant l’audience, comme l’identité et la fonction de certains des acteurs (demandeur, défendeur, avocat, procureur, juges, greffiers), mais pas de tous (jury, témoins). Par ailleurs, l’on pourra avoir accès au chef d’inculpation, à des croquis d’audience, à des extraits des minutes du greffe, aux comptes-rendus journalistiques radiophoniques ou télévisées, aux déclarations d’avocats, etc. Comment alors réconcilier l’ensemble de ces données de natures différentes (écrits, dessins, voix, photographies, vidéos) pour obtenir une vision cohérente du déroulé ? Il faudrait pour cela disposer d’outils « intelligents », capables d’exploiter ces éléments d’information fragmentaires et complémentaires.
Des IA performantes mais souvent restreintes à une seule modalité : son, image, etc.
Des outils intelligents, il en existe qui défraient depuis peu la chronique et sont désormais connus du grand public : ChatGPT, Siri, MidJourney, Dall-E, Google Bard, Mubert, Bing AI,… Leur tactique : exploiter une modalité particulière de données disponibles en très grande quantité sur l’Internet, qu’il s’agisse de documents textuels, de voix enregistrées, d’ensembles d’images ou encore d’extraits vidéo, etc. Des algorithmes dits « d’intelligence artificielle » y sont déployés pour concevoir des modèles d’apprentissage répondant à un objectif bien spécifique : générer du texte ou des images, classifier, etc. Pour y parvenir, ces algorithmes s’efforcent de capturer les briques de base constitutives des données. Ces briques sont abstraites et ne sont souvent pas interprétables par un être humain. Cependant, leur recombinaison ultérieure permet d’imiter certaines tâches, dites intelligentes, que les espèces conscientes savent produire dans le huis-clos de leurs systèmes nerveux : par exemple répondre à des questions écrites, donner l’illusion d’une conversation, créer des images réalistes à partir d’une simple description, truquer une vidéo.
Une intelligence idéale doit exploiter plusieurs modalités
Pour aller plus avant, il semble naturel de chercher à utiliser conjointement différentes modalités disponibles pour les données d’apprentissage. Cela présente bien entendu d’énormes défis, depuis le volume accru de données jusqu’à la combinatoire des associations pertinentes. Des travaux émergent à ce sujet, comme le SeamlessM4T (Massively Multilingual & Multimodal Machine Translation) développé par Meta (ex-Facebook) pour intégrer vision et langage. Pour revenir à notre salle d’audiences, de tels outils pourraient assembler un puzzle multimodal du procès, à partir de toutes les données partielles disponibles, de l’annonce textuelle du verdict par les médias au langage non verbal des acteurs face aux caméras, au prononcé du verdict.
Un exemple de multimodalité : les données omiques
Loin du prétoire, IFPEN rencontre des questionnements analogues. Cependant, ceux-ci concernent des données scientifiques fragmentées et complémentaires, qu’il est souhaitable de soumettre au traitement des outils intelligents. Mais dans quel but, et pour comprendre quel verdict ?
Dans un contexte biologique particulier qui participe à la transition énergétique, IFPEN conduit des recherches dans le domaine de la chimie biosourcée et des biocarburants pour optimiser certains procédés biotechnologiques. Ces derniers reposent sur l’utilisation de micro-organismes dont il est crucial de mieux comprendre le comportement pour en optimiser l’efficacité. Ces connaissances nouvelles permettraient en effet d’améliorer les performances de ces micro-organismes, d’augmenter les rendements de ces procédés biotechnologiques, d’en réduire le coût et d’en favoriser le déploiement.
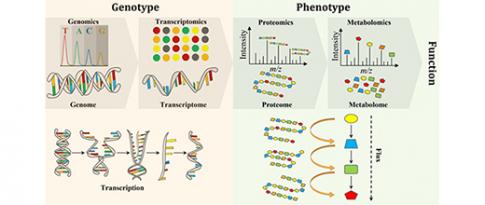
De la cellule, on peut connaître une partie des gènes et de leurs fonctions, ainsi que certaines de leurs actions et interactions. Et l’on peut également accéder à d’autres niveaux d’information importants par la collecte de données dites « omiques » : la génomique pour l’étude des gènes, la transcriptomique pour mesurer l’expression des gènes, l’épigénétique pour comprendre des phénomènes qui ne dépendent pas directement des gènes mais ont une influence notable sur le fonctionnement du micro-organisme. Comme pour le tribunal, ces données omiques éparses fournissent chacune une vue partielle des phénomènes moléculaires à l’œuvre dans une « cellule » et aboutissant à la production de protéines.
IFPEN et CentraleSupélec/INRIA mènent l’enquête avec un travail de thèse
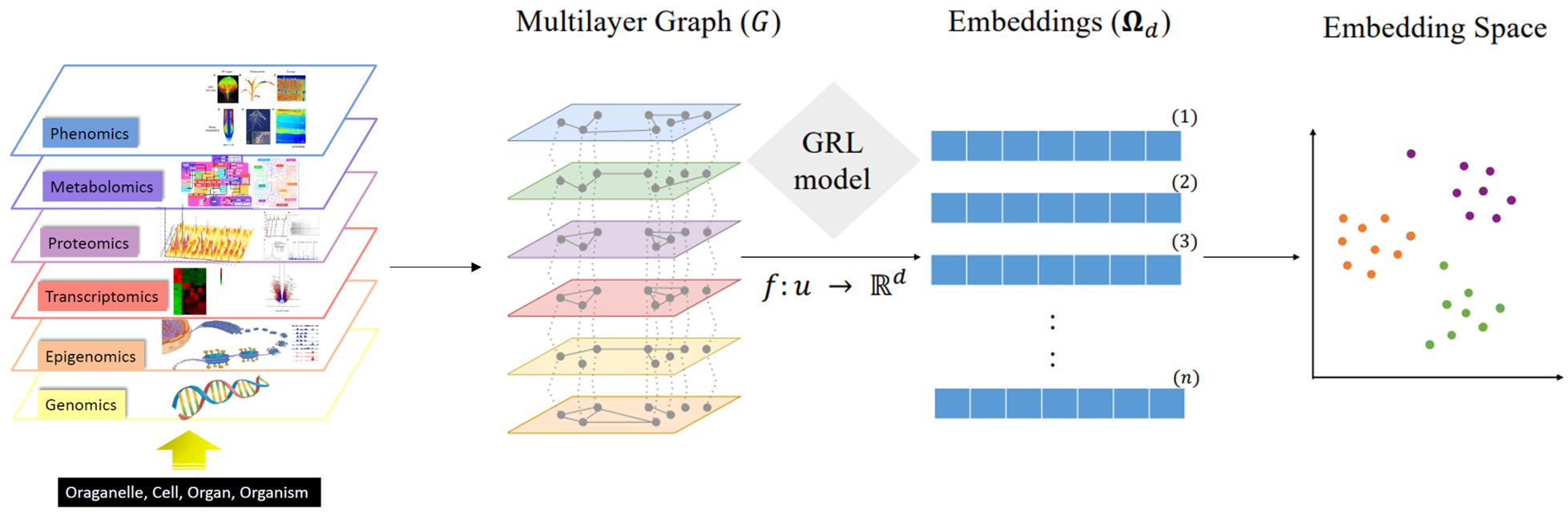
Des experts d’IFPEN et de l’équipe-projet OPIS à CentraleSupélec/INRIA ont collaboré, au travers d’une thèse de doctorat, à la quête de nouvelles méthodes d’analyse du comportement de micro-organismes [1]. Une approche systémique (figure 1) a été adoptée pour prendre en compte différentes modalités de données omiques : génomique, transcriptomique, métabolomique, épigénome, etc. Bien que de nature hétérogène, ces données peuvent être représentées sous forme de graphes. Les réseaux d’interactions résultant, eux-mêmes en interactions entre eux, portent des informations complémentaires qui sont utiles pour la compréhension des mécanismes biologiques sous-jacents. Deux techniques principales d’apprentissage automatique sur des graphes de grande dimension ont été développées, respectivement baptisées BRANEnet [2] et BRANEmf [3]. Ces techniques adaptées à l’analyse des données multi-omiques sont inspirées des méthodes numériques récentes d’analyse de texte ou de réseaux sociaux. Elles mettent en œuvre aussi bien des méthodes statistiques et algébriques que d’apprentissage automatique. Aussi nommées méthodes de plongement (ou embeddings), elles transforment les graphes en nouvelles représentations numériques de dimension réduite, plus faciles à manipuler par un algorithme. Leur application sur les graphes composés de différentes modalités omiques permet alors de capturer l’essentiel des relations entre les gènes. Cette description simplifiée du fonctionnement d’une cellule sert ensuite à effectuer différentes tâches d’intérêt pour la compréhension des phénomènes biologiques : regroupement de gènes liés à la même fonction moléculaire, prédiction de l’interaction de molécules, élucidation de dépendances entre gènes, etc.
Un cas d’étude : la levure de bière
Les méthodes de plongement ont été validées sur des organismes biologiques modèles bien connus comme Saccharomyces cerevisiae, la fameuse levure de bière, par comparaison avec l’état de l’art. Elles fournissent ainsi de nouvelles pistes de compréhension des mécanismes moléculaires pour pouvoir demain améliorer la production de molécules et de produits biosourcés.
Des algorithmes et des données en libre accès
Dans une préoccupation de partage et de science ouverte, l’ensemble des données, les algorithmes et les codes informatiques associées à ces travaux sont mis à disposition du public et de la communauté scientifique, en libre accès1.
1 Codes associés:
- BraneNET
- BraneMF
Références :
[1] Multilayer Graph Embeddings for Omics Data Integration in Bioinformatics, Surabhi Jagtap, Thèse de doctorat en Informatique mathématique, soutenue le 02 février 2023, Université Paris-Saclay.
[2] BRANEnet: Embedding Multilayer Networks for Omics Data Integration. Surabhi Jagtap, Aurélie Pirayre, Frédérique Bidard, Laurent Duval, Fragkiskos D. Malliaros, BMC Bioinformatics, 2022
[3] BraneMF: Integration of Biological Networks for Functional Analysis of Proteins. Surabhi Jagtap, Abdulkadir Çelikkanat, Aurélie Pirayre, Frédérique Bidard, Laurent Duval, Fragkiskos D. Malliaros, Bioinformatics, 2022
Contacts scientifiques : Aurélie Chataignon Pirayre, Laurent Duval, Frédérique Bidard (IFPEN), Fragkiskos Malliaros (OPIS)
Vous serez aussi intéressé par
‘‘BRANE Power’’ : gènes et algorithmes, une alliance pour la chimie verte
THÈSE D’AURÉLIE PIRAYRE, PRIX YVES CHAUVIN 2018
Préparer le changement d’échelle pour des enzymes bien agitées
Les « omiques », sept mercenaires au service de la biotechnologie
Avant leur mise en œuvre dans les procédés de production de biocarburants ou de molécules chimiques biosourcées, les micro-organismes impliqués doivent être optimisés. Cela passe par de l’ingénierie génétique qui consiste à inactiver ou ajouter un ou plusieurs gènes afin d’améliorer l’aptitude à la production de molécules cibles. (...) À IFPEN, le recours à la génomique (ADN) a contribué à rassembler des données sur la composition et la structure des génomes des micro-organismes...